使いたいときにHPCが使えない──研究者の悩み、クラウドで解決できたワケ ゲノム解析の現場を救ったAWS活用(2/2 ページ)
一方でコストが課題に 糸口は「スポットインスタンス」
AWS ParallelClusterの導入作業を始めた当初は、AWSの導入・運用支援やコンサルティングサービスを手掛けるクラスメソッドの支援を仰いだ。同社のエンジニアからAWS ParallelClusterの利用方法についてレクチャーを受け、やがては自分たちだけでも使いこなせるようになったという。
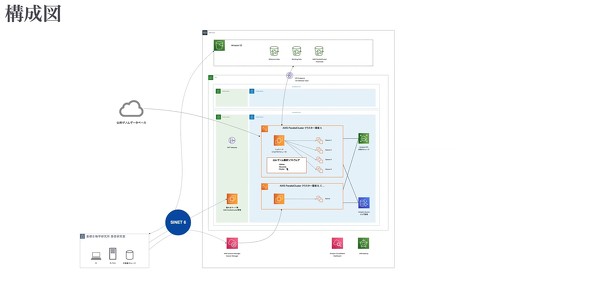
AWS ParallelClusterを使ったゲノム解析処理の全体の流れは以下の通りだ。まずは研究室に設置されているPC端末やLinuxサーバ、HPC環境などから、SINET(学術情報ネットワーク)を通じてAWSに接続。常時起動してある管理サーバのインスタンスにログインし、これまでオンプレミス環境で行っていたのと同じようにジョブスケジューラに計算処理のジョブを投入する。後はジョブスケジューラが、EC2インスタンス(仮想サーバ)によって構成される各クラスタノードにジョブを割り当てて、分散処理を自動実行してくれる。
計算処理中に利用される共用ストレージとしては「Amazon EFS」を利用。一方、計算結果やゲノム情報の生データを保管しておく領域にはより安価な「Amazon S3」を採用し、ストレージコストの最適化を図っている。
ただ、導入に当たっては課題もあった。AWSの利用コストだ。ゲノム解析にはさまざまな種類のソフトウェアを使い分ける必要があり、中には大量のディスクI/Oを発生させるものもある。場合によっては、高いI/O性能を持つ高価なEC2インスタンスでジョブを実行する必要も出てくる。
そこで基礎生物学研究所は、途中で利用を中断される可能性がある代わりに、その時点で空いているEC2のリソースを安く利用できる「スポットインスタンス」を使うことにした。これにより、コストも低く抑えられたという。
「コンピューティングノードには基本的に全てスポットインスタンスを適用している。またAWS ParallelClusterはクラスタの規模を自動的に拡大・縮小してくれる『オートスケール』機能を備えており、常に必要な分だけのノードだけが起動しているので、無駄なコストは発生せず、リソース不足に陥る心配もない。オンプレミスのHPC環境では実現できない機能だと思う」(重信氏)
本業の研究活動に時間を割けるように 一方でメリット最大化には壁も
AWS ParallelClusterの活用により、クラスタを占有でき、オンプレミスのような待ち時間が発生しないHPC環境を手に入れた基礎生物学研究所。コンピュータの運用やメンテナンスの作業などからも解放されたので、本業である研究活動により多くの時間を割けるようになったと重信氏は話す。
HPC環境をクラウド化したメリットは他にもあった。研究者が自らAWS ParallelClusterを使って生成したHPC環境上では、管理者権限を用いて理想の計算環境を自由にスクラップ&ビルドできる。これにより、オンプレミス環境を他の研究者とシェアする場合より、高い自由度で計算環境を構築できるようになったという。
HPCをクラウド化するメリットは、基礎生物学研究所以外の研究機関にもあるだろうと重信氏。例えばコスト面では、クラウドサービスは利用した分のコストのみを負担すればよく、予算不足の大学や研究機関でもHPCを導入しやすくなる。
利用手続きに手間が掛かる問題も解決しやすい。多くの大学や研究機関では、オンプレミスの高価なHPC環境を導入するには煩雑な申請手続きを経る必要があり、導入期間も長期に渡ることが多い。一方、AWS ParallelClusterのようなクラウドサービスならこうした手間や時間を省くことができ、研究者はより本業に専念できると重信氏は話す。
ただし重信氏によれば、クラウド化のメリットを享受するには、ある程度の知識やスキルも求められるという。
「AWS ParallelClusterを効率的に利用するには、利用するアプリケーションの特性に見合ったサービスを見極めて選ぶ必要があり、どうしてもAWSの知識やスキルが必要になる。今回の導入ではクラスメソッドに伴走型のサポートをお願いしてスキル不足を補ってもらったが、やはり適切に使いこなすためにはユーザー側の『AWS力』を高める必要があると感じた」
関連記事
 「正直バブリー」 地方の中小企業がAWS導入でビジネスチャンスを逃さなかった話
「正直バブリー」 地方の中小企業がAWS導入でビジネスチャンスを逃さなかった話
「注文数も客単価も増え、正直バブリー」──AWSを導入していたことで、突然のビジネスチャンスを逃さず済んだ地方中小。いきなりのチャンスをどうつかんだか、キーパーソンに聞く。 「すずめの戸締まり」制作でもAWSが活躍 レンダリングで活用 エンドロールにも……
「すずめの戸締まり」制作でもAWSが活躍 レンダリングで活用 エンドロールにも……
11月11日に公開となった新海誠監督の映画最新作「すずめの戸締り」。制作に当たってはレンダリングなどの用途にクラウドサービス「Amazon Web Services」を活用したという。エンドロールにもロゴが……。 「職員室でしかPCが使えない」 “生徒1人にPC1台”の裏で、進まぬ教育現場のデジタル化 意識改革が必要なのは誰なのか
「職員室でしかPCが使えない」 “生徒1人にPC1台”の裏で、進まぬ教育現場のデジタル化 意識改革が必要なのは誰なのか
“生徒1人にPC1台”施策の裏で、まだ進まない教育現場のIT活用。実現に当たって意識改革が必要なのは教師か、教育委員会か、自治体か。問題の本質を教育現場や有識者への取材から探る。 「誰でも使えるシステムがすぐ必要だった」 オンライン授業迫られた文系大学の奔走 タイムリミットは2週間
「誰でも使えるシステムがすぐ必要だった」 オンライン授業迫られた文系大学の奔走 タイムリミットは2週間
新型コロナウイルスの影響を受け、オンライン授業の実施を迫られた小樽商科大学。教員内のITリテラシーに格差があり、シンプルで安価な配信システムを2週間で構築する必要があった。 「量子超越性」を持つ光量子コンピュータ、AWSで利用可能に スパコン富岳で9000年かかる計算を36マイクロ秒で
「量子超越性」を持つ光量子コンピュータ、AWSで利用可能に スパコン富岳で9000年かかる計算を36マイクロ秒で
カナダの量子ベンチャーXanadu(ザナドゥ)は、特定のタスクで世界最高性能のスーパーコンピュータの計算速度を上回るとする光量子コンピュータ「Borealis」をAmazon Web Services(AWS)上で提供すると発表した。
関連リンク
Copyright © ITmedia, Inc. All Rights Reserved.
Special
PR