東大医科研とIBMが新型コロナウイルスのゲノム解析システムを共同開発 変異株対策に一手
» 2021年06月09日 18時42分 公開
[松浦立樹,ITmedia]
東京大学医科学研究所付属ヒトゲノム解析センターと日本アイ・ビー・エムは6月8日、新型コロナウイルスの変異状況や流入経路を迅速に解析できるシステムを開発し、運用を始めたと発表した。
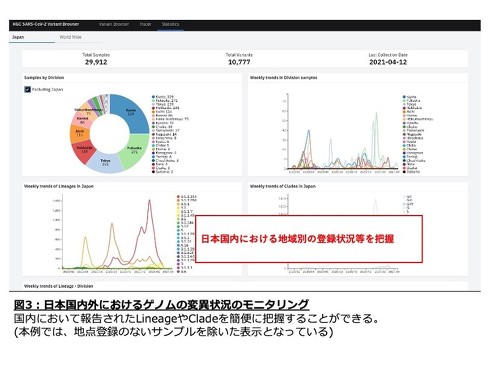
開発したシステム「HGC SARS-CoV-2 Variant Browser」は、新型コロナウイルスのゲノム情報を解析することで、「どういった変異を持っており」「いつどの国から流入し」「どのように感染してきたのか」といった情報を把握できるという。
ヒトゲノム解析センターによれば、5月時点で世界中の新型コロナウイルス感染者170万人以上のゲノム情報が「GISAID」と呼ばれる研究機関向けのデータベースに公開されている。しかし、このサンプル数を迅速に解析する体制が十分に整備されていなかった。
今回開発したシステムを活用することで、新たな変異株のいち早い発見が期待できる他、感染経路の推定によって、感染リスクが高い場所や行動の推測にもつながるという。
HGC SARS-CoV-2 Variant Browserは、IBM内の開発チームが手掛けた「SARS-CoV-2 Variant Annotator」と「SARS-CoV-2 Variant Browser」の技術をベースに開発。ヒトゲノム解析センターが保有するスーパーコンピュータ「SHIROKANE」で稼働している。
関連記事
 政府が「ワクチン接種状況ダッシュボード」公開 性別や都道府県別に可視化
政府が「ワクチン接種状況ダッシュボード」公開 性別や都道府県別に可視化
政府が、全国の新型コロナワクチンの接種状況を一覧にまとめた「ワクチン接種状況ダッシュボード」を公開。統計情報をまとめたCSVやJSON形式のファイルも提供する。 IBM、研究用量子コンピュータを東大キャンパス内に設置 研究施設もオープン
IBM、研究用量子コンピュータを東大キャンパス内に設置 研究施設もオープン
日本アイ・ビー・エムが、東京大学浅野キャンパス(東京都文京区)に量子コンピュータ技術のハードウェアに関する研究や開発を行うテストセンターを開設したと発表。日本国内での研究開発の促進や人材育成を狙う。 NEC、本社など4カ所で社員1万人にワクチン優先接種へ 政府の“職域接種”に協力
NEC、本社など4カ所で社員1万人にワクチン優先接種へ 政府の“職域接種”に協力
NECはグループ社員や協力会社の一部の社員、計1万人を対象に新型コロナワクチンを優先的に接種すると発表した。本社(東京都港区)や関西支社(大阪市中央区)、玉川事業場(神奈川県川崎市)、府中事業場(東京都府中市)の会議室などで、常駐する産業医や看護師が社員に接種する。 ゲノム情報を暗号化したまま解析、プライバシーを保護 NECと阪大が実験
ゲノム情報を暗号化したまま解析、プライバシーを保護 NECと阪大が実験
NECと大阪大学が、データを暗号化したまま処理できる技術「秘密計算」をゲノム解析システムに適用する実験を行い、実用性を示した。 東芝、盗聴不可能な「量子暗号」でヒトゲノム約500GBの伝送に成功 世界初
東芝、盗聴不可能な「量子暗号」でヒトゲノム約500GBの伝送に成功 世界初
東芝と東北大学は1月14日、「量子暗号通信」を用いて、人のゲノムデータ約500GBを約7キロ離れた施設へ伝送することに世界で初めて成功したと発表した。同社は近く、量子暗号通信で事業展開を始める見込み。
関連リンク
Copyright © ITmedia, Inc. All Rights Reserved.
Special
PRアイティメディアからのお知らせ
SpecialPR
あなたにおすすめの記事PR